Exames > Centogene > Sequenciamento Completo de Exoma
Sequenciamento Completo de Exoma
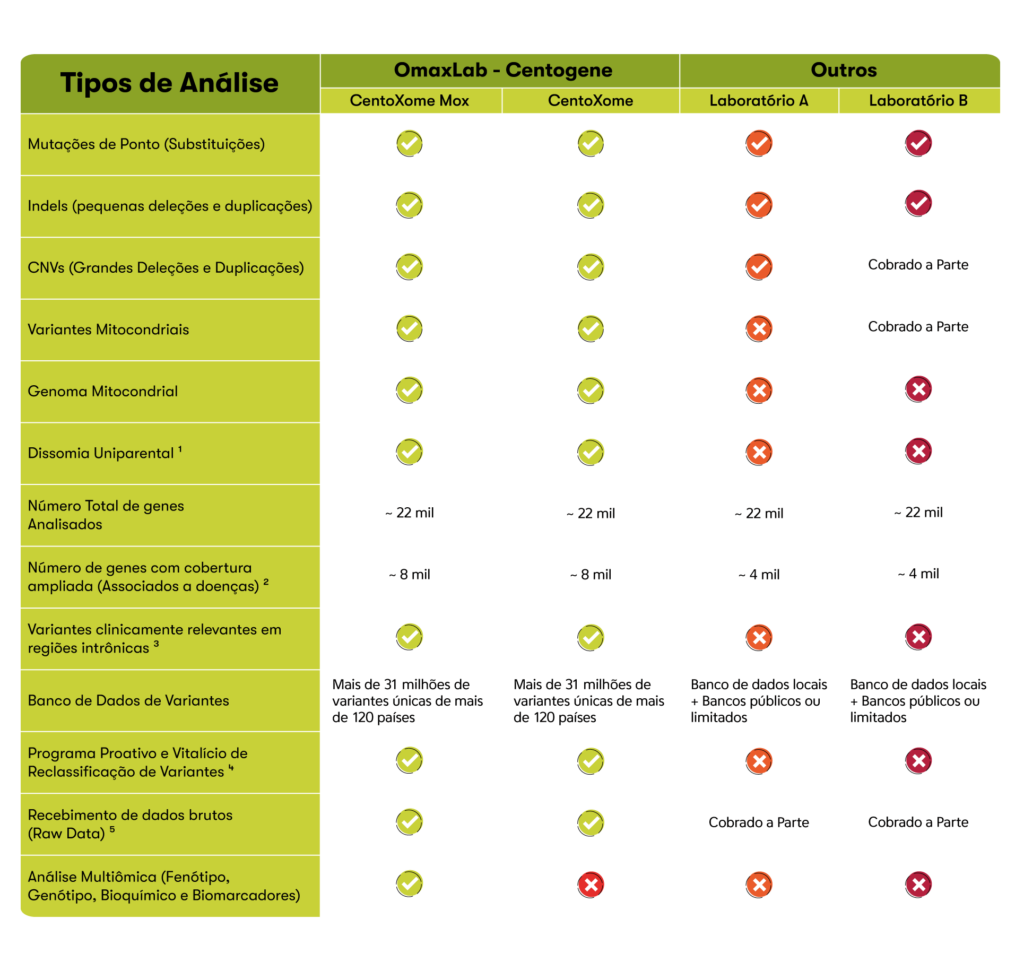
CentoXome® e CentoXome® MOx
O que é o Sequenciamento Completo de Exoma?
É um exame genético abrangente que é capaz de detectar alterações no DNA em aproximadamente 20.000 genes de um paciente. Ao focar em todas as regiões codificadoras de proteínas do genoma – o exoma – o CentoXome oferece a cobertura necessária para diagnosticar pacientes de forma rápida e confiável.
Quando é recomendado realizar o exame?
O exoma pode ser realizado nos seguintes casos:
• Pacientes que apresentam fenótipos heterogêneos complexos que são sugestivos de várias condições, tornando os painéis de grupos de genes a melhor opção;
• Casos nos quais o teste genético anterior não foi bem-sucedido. Por exemplo, paciente com atraso no desenvolvimento neurológico, com irmãos afetados de forma semelhante e teste negativo com microarrays;
• Sintomas ou histórico familiar de um paciente sugerem uma causa genética das doenças.
CentoXome®
O sequenciamento completo do exoma (WES), CentoXome®, é um teste genético abrangente que identifica alterações no DNA de um paciente que são causais ou relacionadas a suas preocupações médicas. Ao focar em todas as regiões codificadoras de proteínas do genoma – o exoma – o WES oferece a cobertura necessária para diagnosticar pacientes de forma rápida e confiável.
CentoXome® MOx
Multiômica representa os subconjuntos de ômicas – genômica, transcriptômica, proteômica, metabolômica e fenômica. Com um quadro clínico completo por meio de dados multiômicos, o processo e a precisão do tratamento de doenças raras estão sendo redefinidos. Somos capazes de obter insights multicamadas para fornecer o diagnóstico mais preciso, desenvolver melhores modelos de doenças e, assim, fornecer medicamentos mais precisos.
Por que escolher o CentoXome®?



Compare e Comprove
O design e o serviço do CentoXome oferecem a qualidade e o desempenho ideais do líder mundial e parceiro confiável em diagnósticos de doenças raras, com excelente cobertura clínica e poder de diagnóstico clínico incomparável em um único teste.
Combinando insights de nosso extenso e exclusivo banco de dados centrado em doenças raras com tecnologia ômica superior, pacientes e médicos se beneficiam de uma abordagem única que aumenta o rendimento diagnóstico em até 20% em comparação com a rotina WES 3-8 por meio de cobertura aprimorada do exoma, genoma mitocondrial completo de genes e variantes clinicamente associados conhecidos.
Informações sobre o Exame
• Profundidade média ≥ 100x
• Cobertura altamente uniforme de todo o exoma (~ 20.000 genes) e genoma mitocondrial (37 genes); ≥ 98,0% regiões-alvo cobertas em ≥ 20x
• Todas as regiões de codificação de proteínas de genes nucleares, limites de exon-íntron de +/- 10 bp e genoma mitocondrial completo.
Cobertura ampliada de regiões clinicamente relevantes
• ~ 8.000 genes associados a doenças; ≥ 99,5% regiões-alvo cobertas em ≥ 20x
• 99% de todas as variantes clinicamente relevantes conhecidas em regiões codificantes e não codificantes extraídas de HGMD®, ClinVar e Bio / Databank da CENTOGENE para doenças raras
Detecção avançada e sensível de quase todos os tipos de variantes em um único teste
• Detecção altamente sensível e específica de SNVs, InDels, CNVs de alterações de nível de éxon a nível citogenômico, UPD* e variantes de mtDNA com heteroplasmia ≥15%
•Sensibilidade SNVs e InDels (≤55pb) > 99,6% CNVs (≥3 éxons) ** >95,0%
• A especificidade de >99,9% é garantida para todas as variantes relatadas ***
* A triagem UPD é realizada usando um algoritmo específico interno para as seguintes regiões cromossômicas clinicamente relevantes bem conhecidas: 6q24, 7, 11p15.5, 14q32, 15q11q13, 20q13 e 20.
** O software de detecção de CNV tem uma sensibilidade >95,0% para todas as deleções homozigóticas/hemizigotas e mitocondriais, bem como deleções/duplicações heterozigóticas e duplicações homozigotas/hemizigotas abrangendo pelo menos três éxons consecutivos
*** Variantes com baixa qualidade e/ou zigosidade incerta são confirmadas por métodos ortogonais (ou seja, SNVs e InDels por sequenciamento de Sanger; CNVs por amplificação de sonda multiplex dependente de ligação, MLPA; reação em cadeia da polimerase quantitativa, qPCR; ou microarray cromossômico, CMA)